DeepST
论文阅读记录
DeepST
- 标题:DeepST: identifying spatial domains in spatial transcriptomics by deep learning
- 作者:Harbin Institute of Technology
- 发表会议/期刊:Nucleic Acids Research
- 年份:2022
- 链接:DeepST
主要内容简介
- Motivation:
- 先前方法主要依赖线性主成分分析来提取基因表达的高变特征,因此无法建模复杂的非线性相互作用
- 未充分利用空间信息,且在预测组织结构方面存在局限
- 大多数分析大量ST数据的空间方法无法正确校正批次效应,且不能处理其他空间组学数据,使其通用性不足
方法与创新点
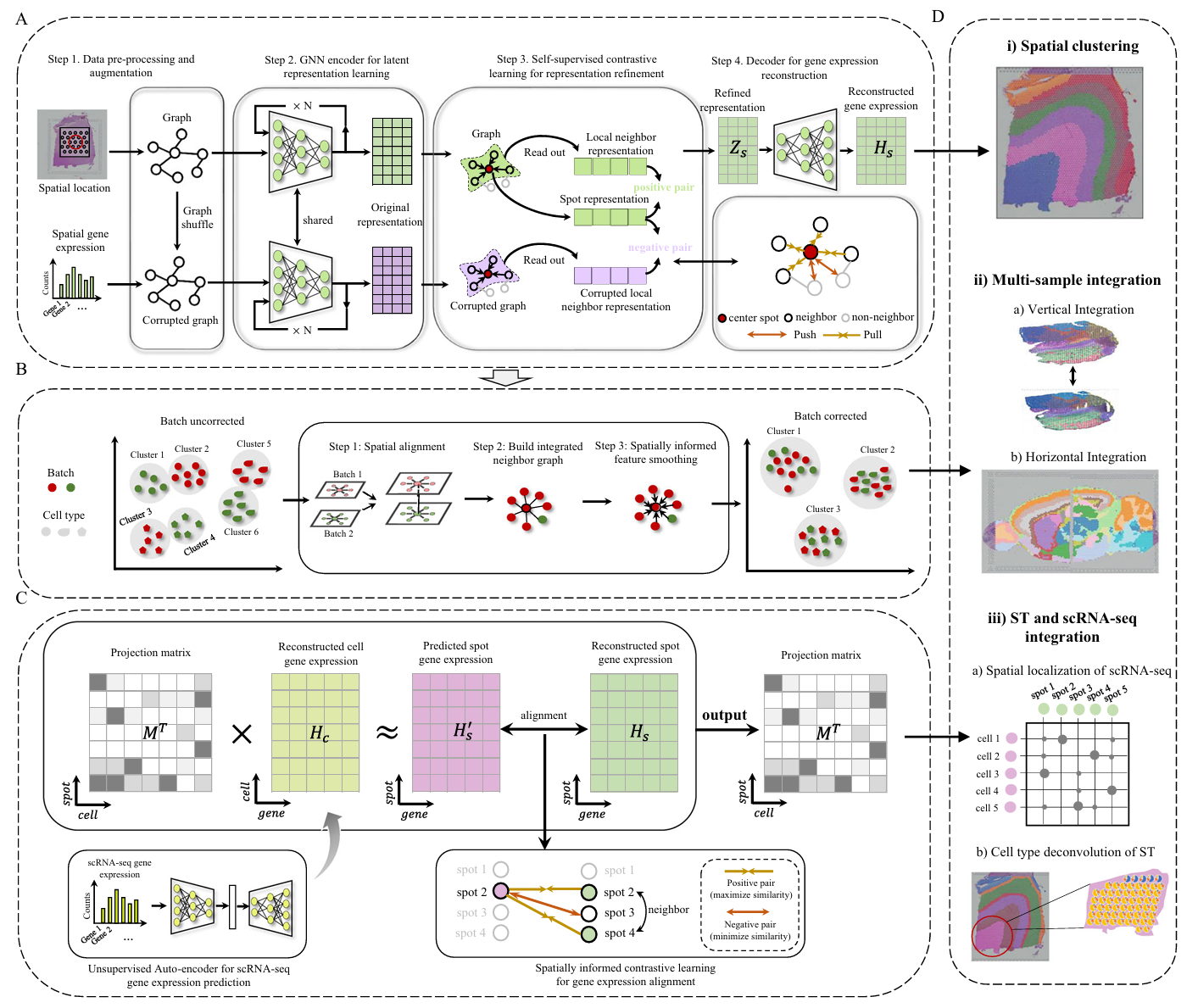
总体框架:
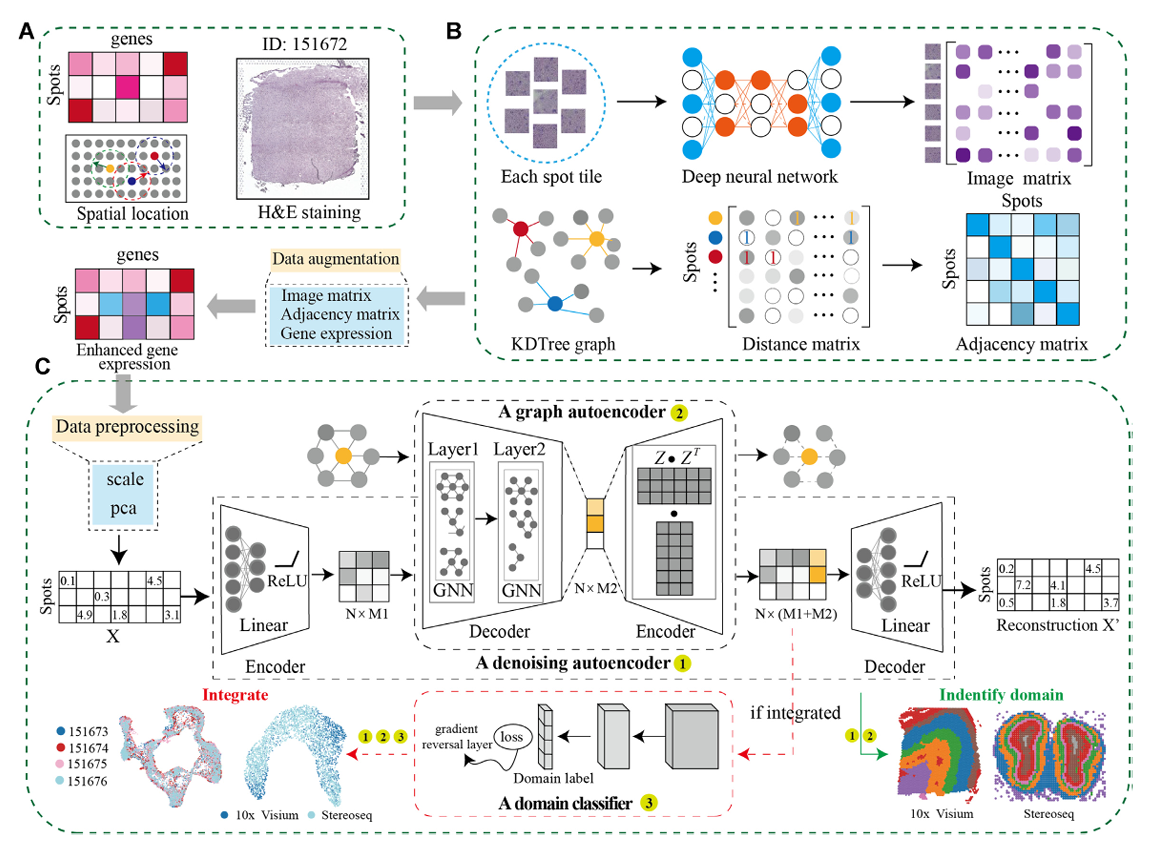
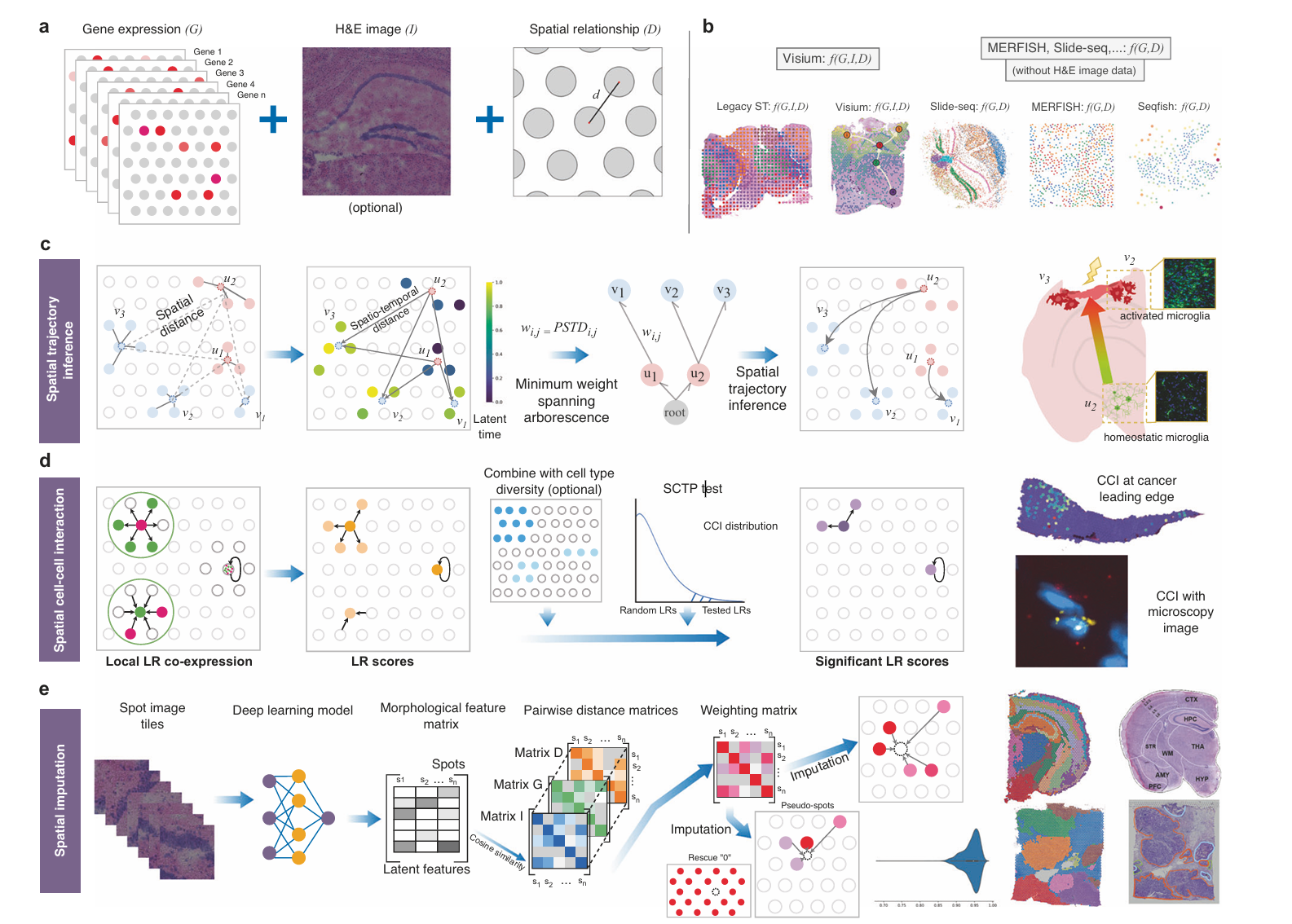
方法概述:
Spatial data augmentation(平滑补偿机制):
- 基因表达相关性(GCij):基于标准化后的余弦距离计算点对间表达谱相似性;
- 形态学相似性(MSij):
- 从H&E图像提取局部区块,经数据增强(旋转、锐化等)后通过预训练CNN(如Inception v3)提取2048维特征;
- 使用PCA降维至50维主成分,最终通过余弦距离量化形态相似性;
- 空间邻近性(SWij):基于坐标计算点间距离,定义半径γ(最近4点距离均值+方差)作为邻居判定阈值。
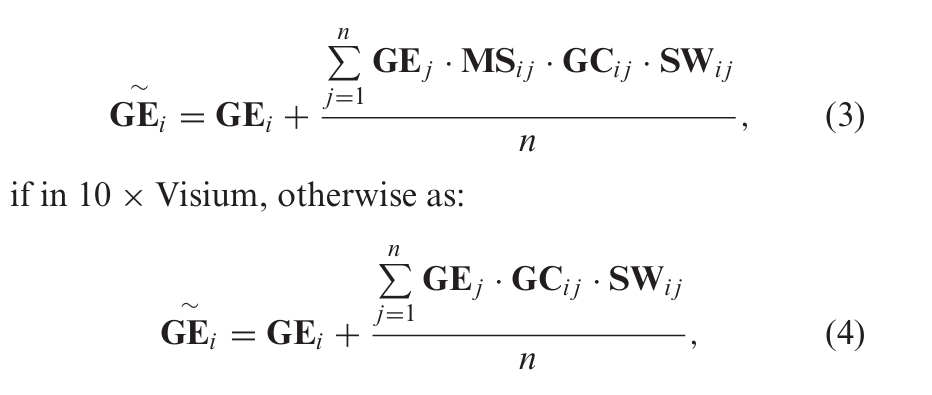
Graph construction:
- 基于空间坐标计算spots间距离,使用BallTree算法,默认top12为邻居,构建Graph
Denoising autoencoder:

Variational graph autoencoder(VAE):

Domain adversarial neural networks:
- 目的:DAN的目的是将不同分布的源域和目标域映射到同一特征空间,使空间中的距离尽可能接近。

本博客所有文章除特别声明外,均采用 CC BY-NC-SA 4.0 许可协议。转载请注明来源 小鱼日记!