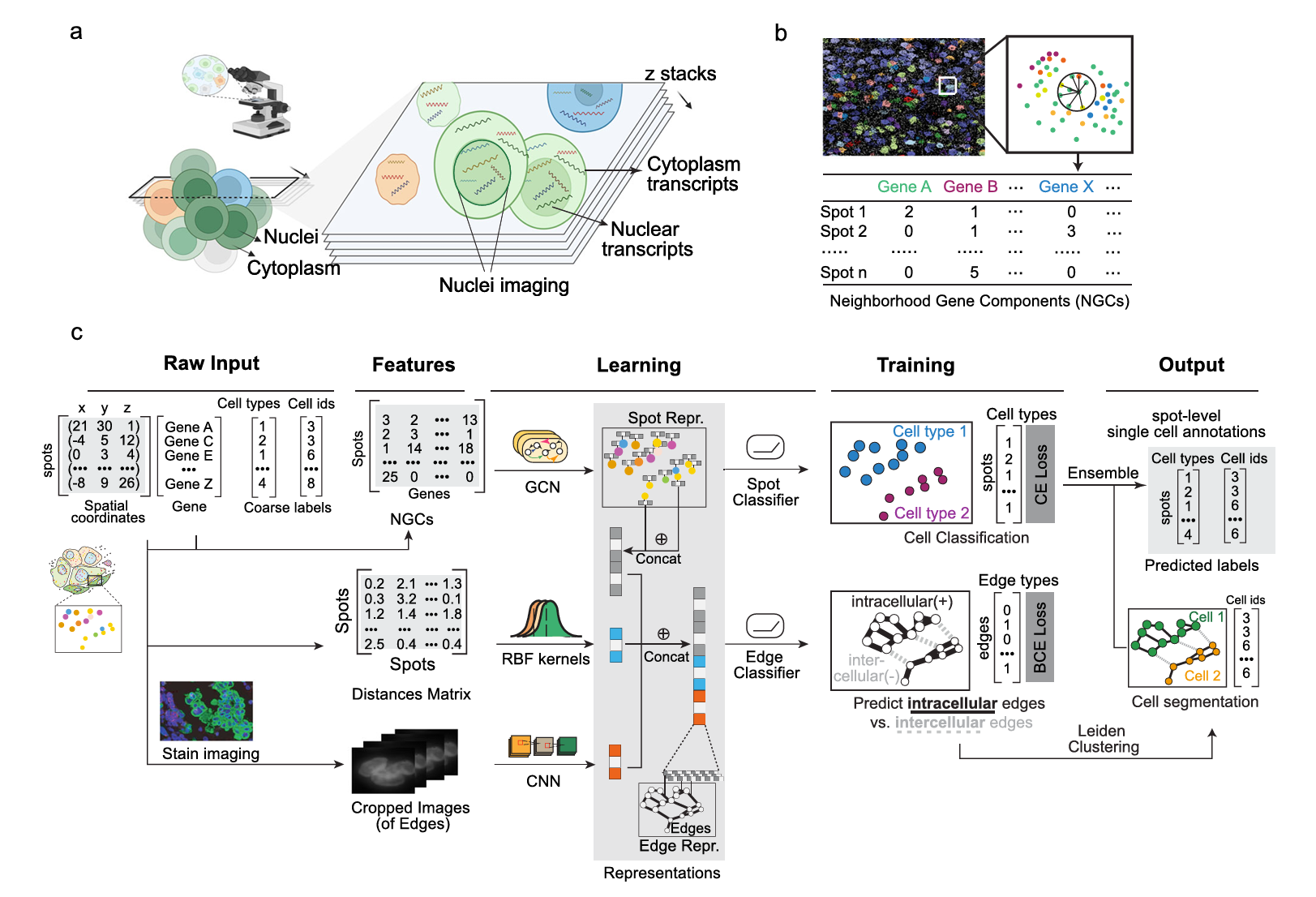
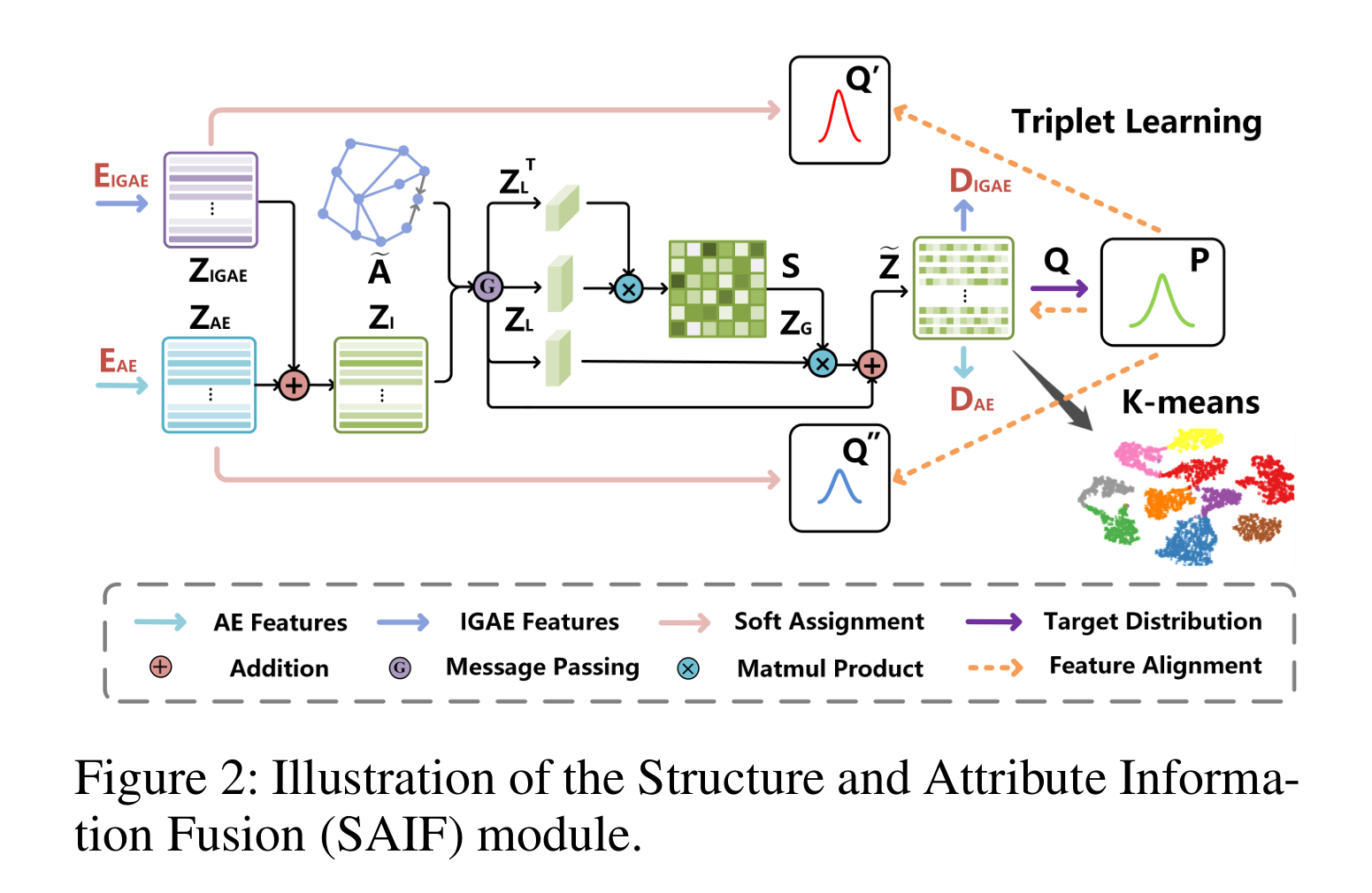
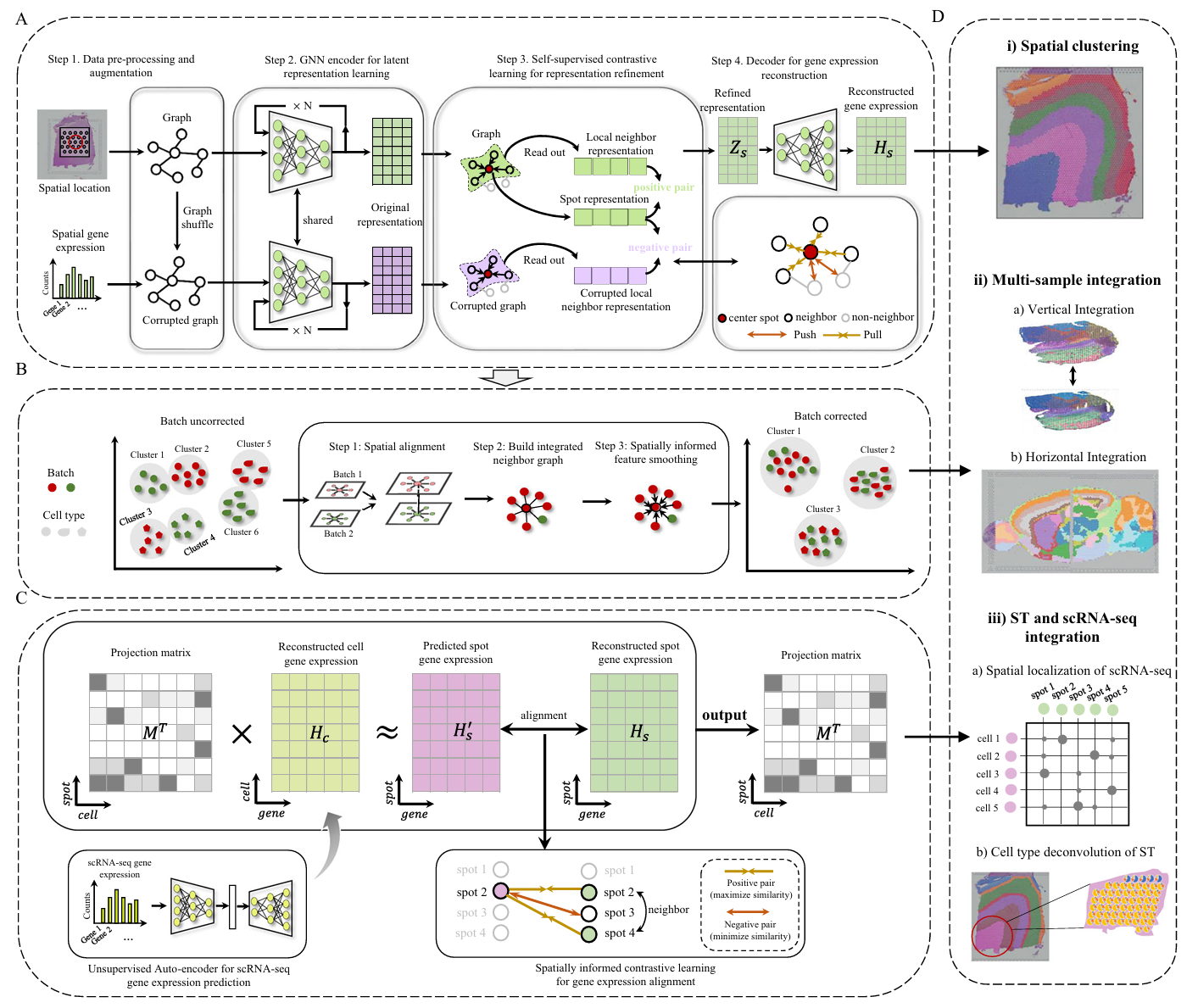
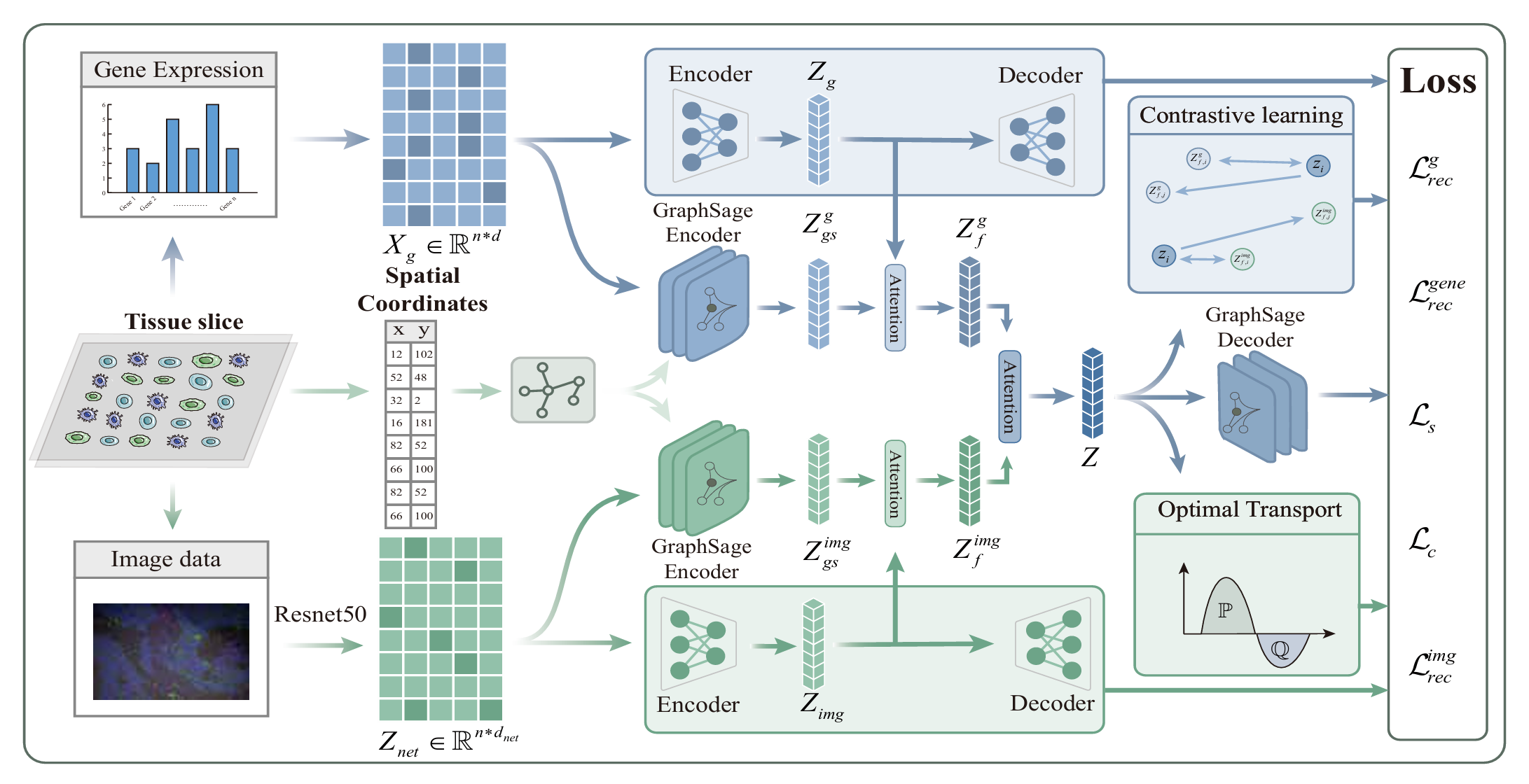
基于代码理解MAFN
📖 论文阅读记录
1. 基本信息
- 论文题目:Multi-View Adaptive Fusion Network for Spatially Resolved Transcriptomics Data Clustering
- 作者/机构:China University of Geosciences
- 会议/期刊:IEEE TRANSACTIONS
- 年份:DECEMBER 2024
- 论文链接:MAFN_IEEE
2. 方法总览
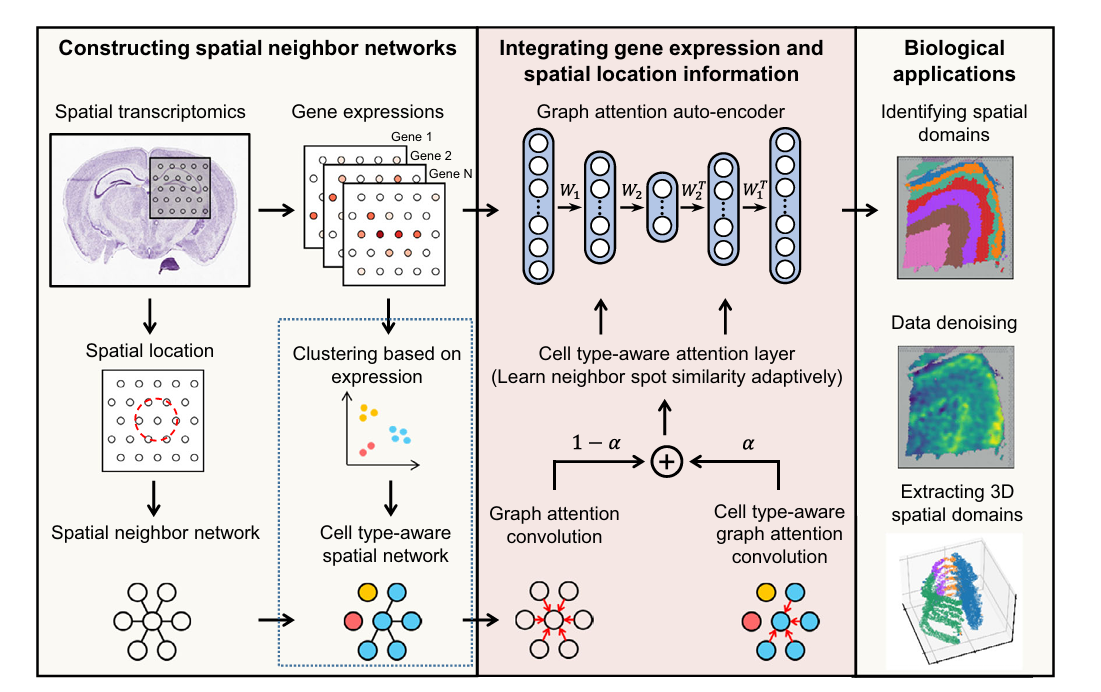
总体框架:
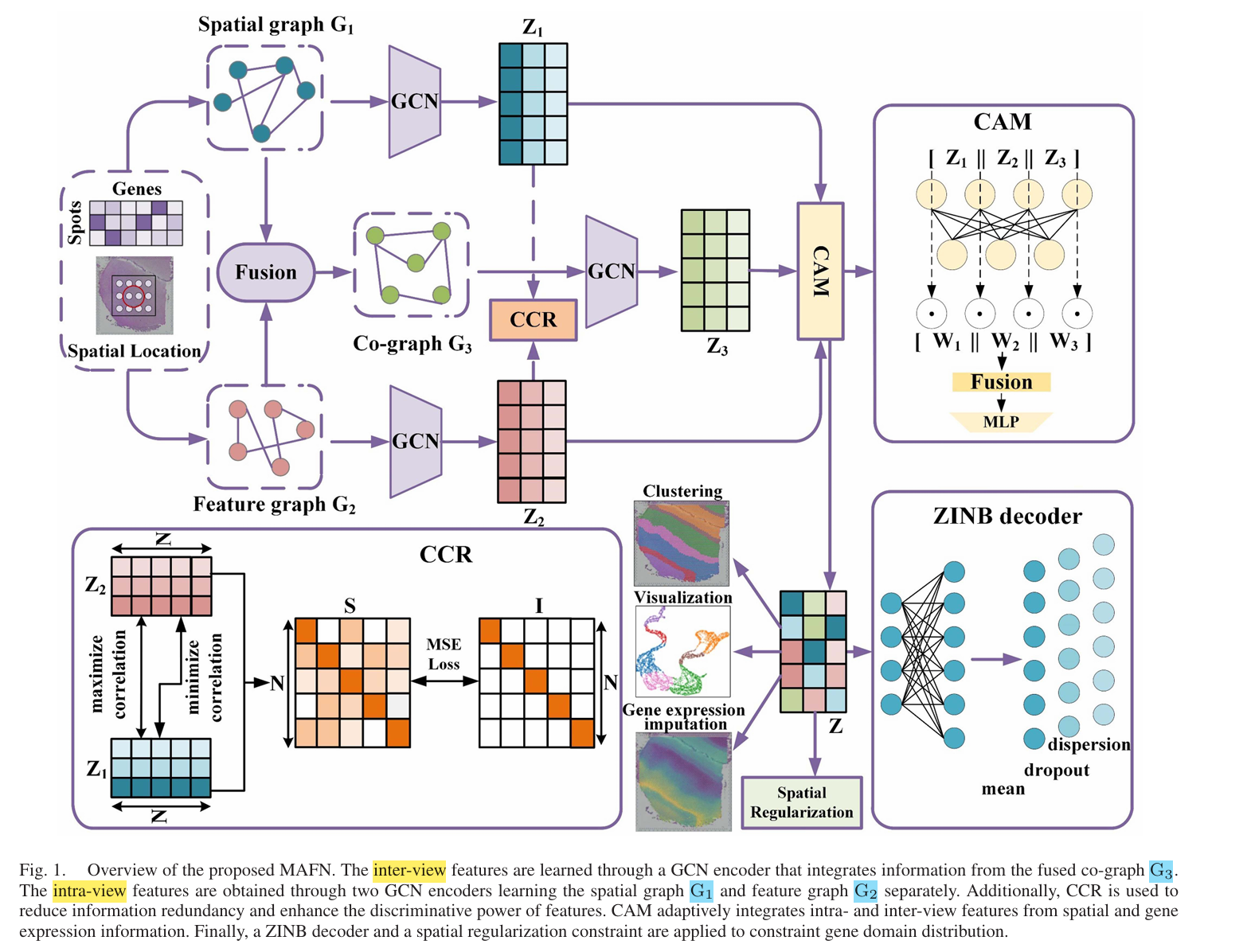
关键技术路线:
- spatial graph G1(euclidean distance & r), feature graph G2(cosine similarity & kNN)
- Inter-View Complementary Features Learning (GCN)
- Intra-View Discriminative Features Learning(GCN + Lc –> (S-I)^2)
- Cross-View Attention Module (CAM)
- Gene Domain Distribution Constraints
正则化损失公式Lr:
$$
L_R = - \sum_{i=1}^N \left( \sum_{j \in R_i} \log(\sigma(H_{ij})) + \sum_{k \notin R_i} \log(1 - \sigma(H_{ik})) \right)
$$ZINB分布损失公式Lz:
$$
L_Z = -\frac{1}{m(n_s + n_t)} \sum_{i=1}^{m} \sum_{j=1}^{n_s + n_t} \ln, p_{ZINB}(x_{ij} \mid b_i)
$$
- Overall Loss Function:
$$
L = \lambda_1 L_C + \lambda_2 L_R + \lambda_3 L_Z
$$
3. 代码细节
- 代码仓库: MAFN_GitHub
- 复现细节(可以用Spatial-MGCN仓库的readme.md作为复现参考):
数据下载:以DLPFC-151507切片为例,下载数据并解压到
data文件夹下(可在Spatial-MGCN仓库中找到数据,官方数据集下载地址我没去找QAQ)运行DLPFC_generate_data.py:对原始数据进行处理,存储为Anndata格式,构建G1和G2(运用utils.py中的函数)
- 知识点:
- AnnData 结构表格
字段 数据类型 维度 / 形状 作用 / 描述 示例 X ndarray / sparse (n_cells × n_genes) 存储每个细胞的基因表达矩阵 原始 counts 或归一化后的表达 obs DataFrame (n_cells × n_obs) 细胞注释信息,每行对应一个细胞 cell_type, batch, cluster_label var DataFrame (n_genes × n_var) 基因注释信息,每行对应一个基因 gene_symbol, highly_variable obsm dict / ndarray (n_cells × k) 细胞低维表示或嵌入向量 PCA, UMAP, tSNE, 自定义 embedding ( mean)varm dict / ndarray (n_genes × k) 基因低维表示或载荷 PCA loadings layers dict / ndarray (n_cells × n_genes) 多层表达矩阵 raw counts, normalized, denoised obsp dict / sparse (n_cells × n_cells) 细胞-细胞稀疏矩阵 kNN 图的 connectivities, distances varp dict / sparse (n_genes × n_genes) 基因-基因稀疏矩阵(可选,较少用) PCA 基因相似性矩阵 uns dict 无固定形状 无结构结果或其他参数 colors, neighbors params, pca info - notice:
- 我将源码里os.mkdir改成了os.makedirs,防止文件缺失报错
- 知识点:
运行DLPFC_test.py:
-note:
- ZINB分布损失,需要三个参数:mean,disp,pi 均是通过models/decoder得到
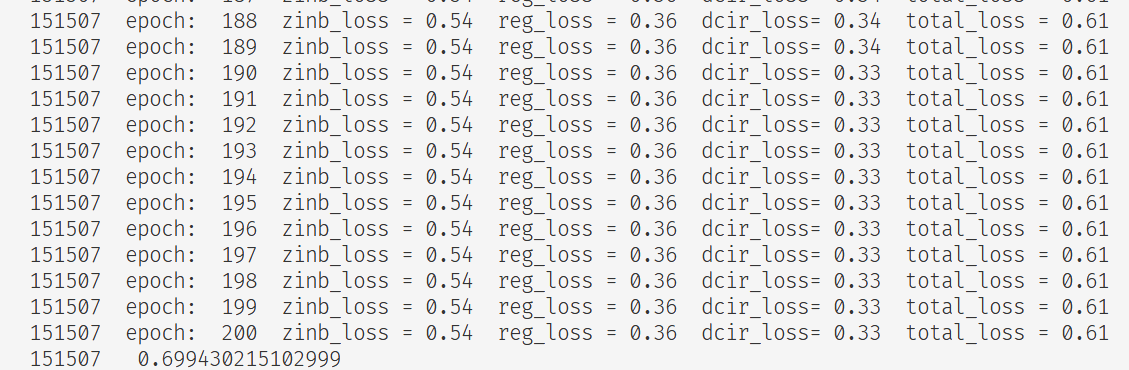
结果:
训练ing:
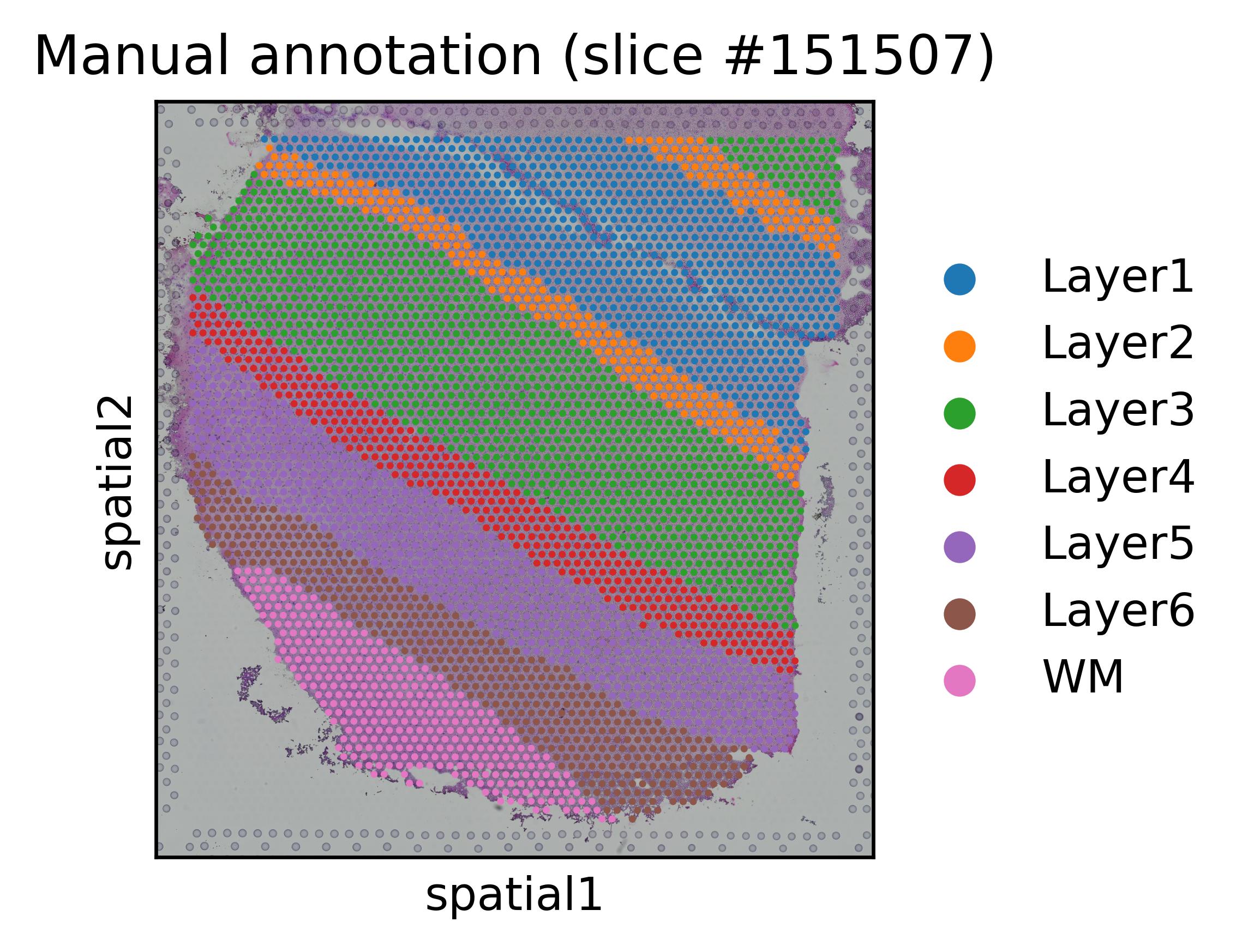
可视化:
- 左图为手动标注的细胞类型,右图为MAFN聚类结果
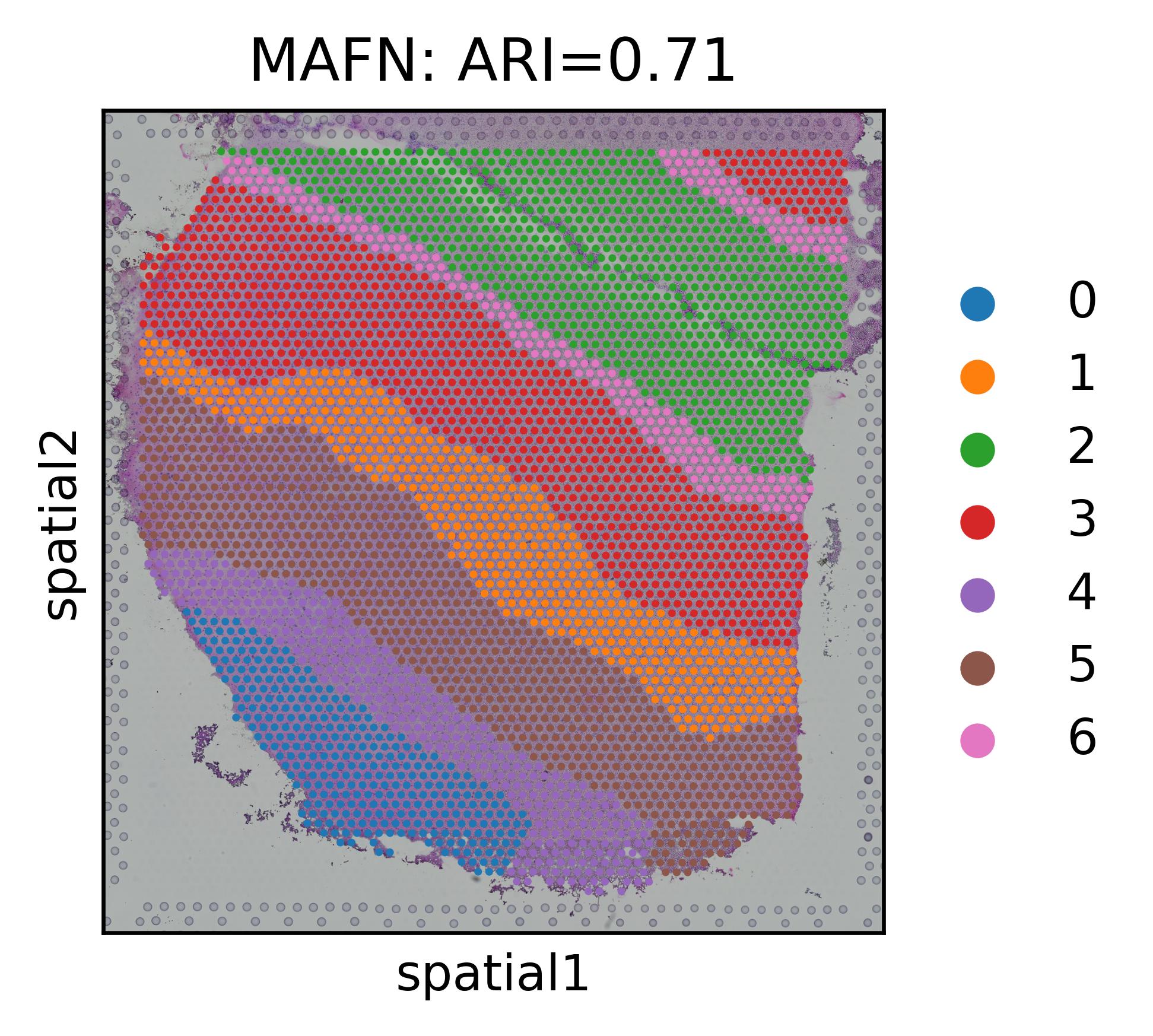
- UMAP和PAGA可视化
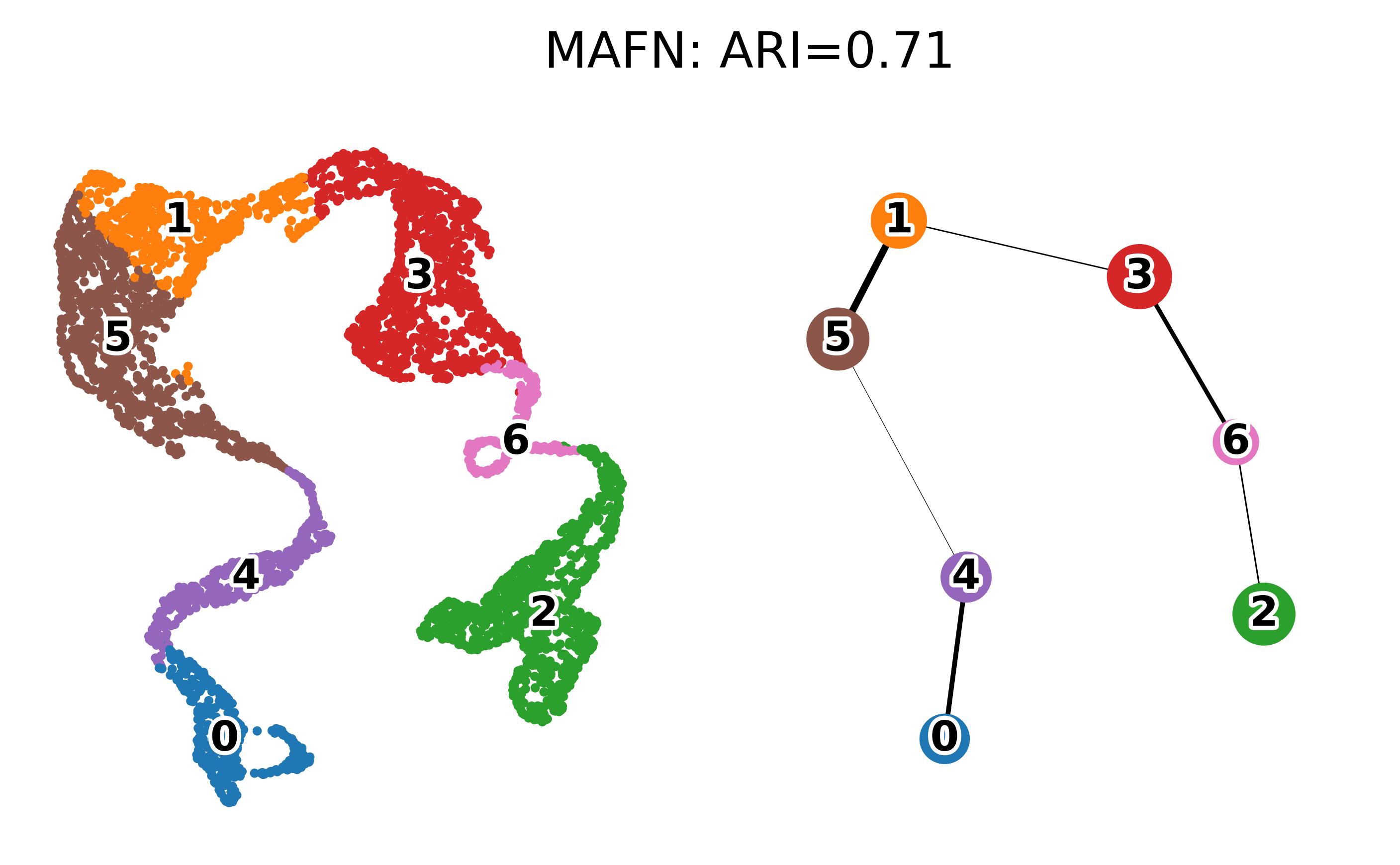
- MAFN聚类标签和embedding representation

4.Summary
- 熟悉Scanpy,单细胞数据集,Anndata数据结构
- umap可视化 and so on…
5. References
本博客所有文章除特别声明外,均采用 CC BY-NC-SA 4.0 许可协议。转载请注明来源 小鱼日记!